本网讯 7月8日,国际权威期刊《自然通讯》(Nature Communications)在线发表了中南大学计算学院王建新教授团队和美国克莱姆森大学罗峰教授、中山大学中山眼科中心肖传乐副研究员合作的最新研究成果“使用太平洋生物科学公司循环共识测序方法检测基因组甲基化位点和分型(DNA 5-methylcytosine detection and methylation phasing using PacBio circular consensus sequencing)”。该论文提出了可准确检测全基因组单倍型5mC的方法和流程,中南大学计算机学院倪鹏、聂藩为论文共同第一作者,王建新教授为论文共同通讯作者,中南大学生命科学学院、湘雅医院李津臣教授团队为本研究提供了重要数据支持,中南大学为第一署名单位。

近年来三代测序技术(牛津纳米孔测序和PacBio单分子实时测序)的迅速发展为基因组学的研究带来了新的机遇。其中,基于单分子实时测序技术,PacBio公司进一步提出了HiFi测序技术,它采用循环共识测序策略进行测序,大大提高了测序读数的准确性。PacBio HiFi测序技术已在基因组组装、SNP检测等方面得到了广泛应用,极大地推动了基因组学的发展。然而由于其测序信噪比相对较低,与纳米孔测序技术相比,PacBio HiFi测序技术并未在DNA甲基化检测方面得到较好的应用。
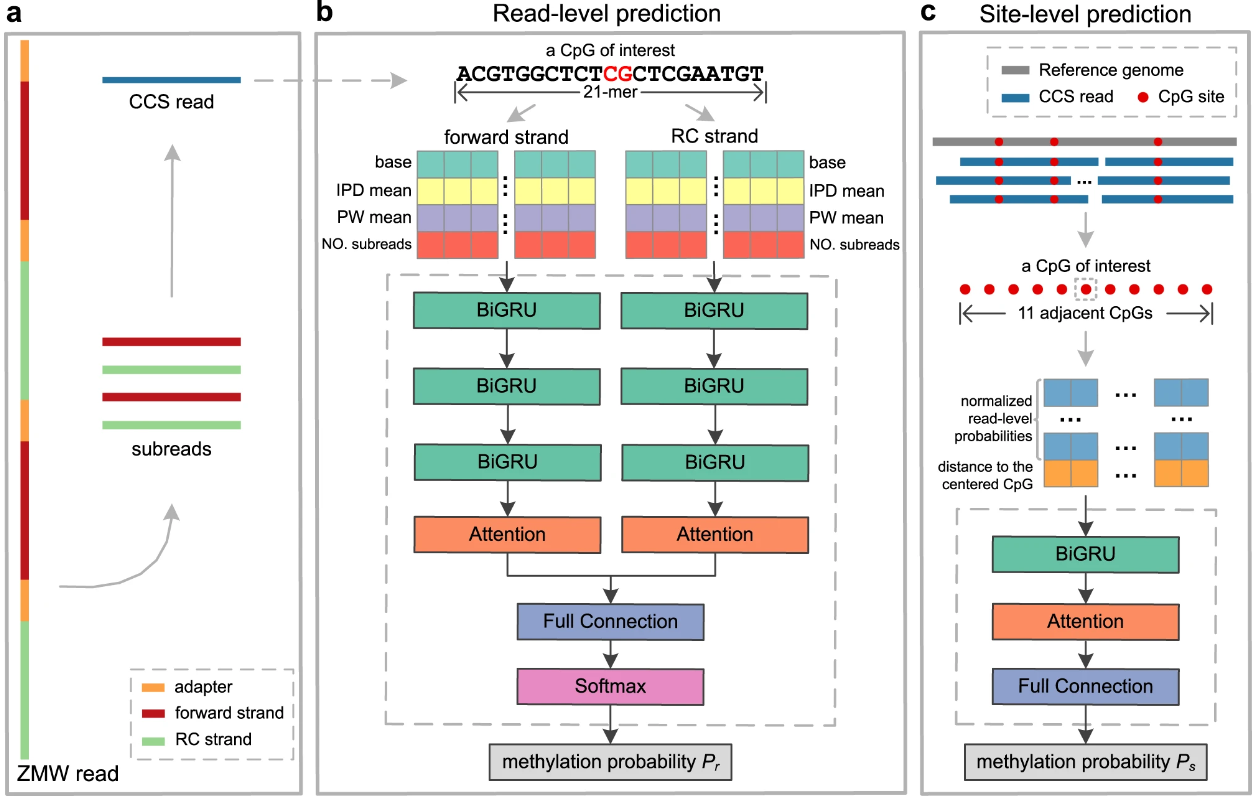
ccsmeth方法原理图
针对这一局限,王建新教授团队的研究论文基于PacBio HiFi测序,结合5mC位点的对称特性和聚集特性,设计深度神经网络,提出了可准确检测全基因组5mC甲基化的方法ccsmeth。ccsmeth是一种使用CCS读数检测DNA 5mCpGs的深度学习方法。ccsmeth在读数水平和基因组位点水平的预测性能均高于已有方法(高>4%),特别在低覆盖度条件下优势明显,可大幅降低检测所需测序成本。本文进一步设计了基于HiFi测序的单倍型5mC甲基化检测流程,并利用Nextflow框架开发了相应工作流ccsmethphase。该研究验证了HiFi可对人类基因组中更多的CpG位点进行甲基化检测和分型,与二代短读数测序相比,其可分型CpG数量是二代测序的3倍以上。
(一审:李贝 二审:韩艳 三审:李殷)
本文链接:https://news.csu.edu.cn/info/1004/156562.htm